
更新时间:2020-04-27
近日,南科大医学院教授张文勇、研究助理教授栾合密等在国际著名生物信息学与计算生物学期刊Bioinformatics上发表题为“CPVA: a web-based metabolomic tool for chromatographic peak visualization and annotation”的论文。该研究创新提出了以色谱中心策略算法(CCS)提高基于质谱的组学数据质量,并搭建了R-Shiny的Web应用CPVA,实现了组学数据在线交互式的处理(图1)。

CPVA是一个交互式网页工具,用于对峰形进行可视化和注释,揭示峰形的隐藏特性,从而提高非目标组学研究的数据质量。有审稿人认为该论文及其工具填补了非靶向组学数据处理过程中的一个主要的漏洞。
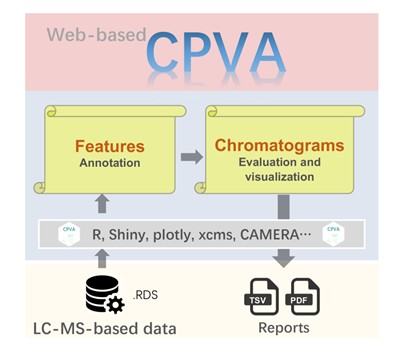
图1 CPVA算法与在线工具的流程示意图
定量系统生物学是描述生命体的细胞、组织、器官以及整体水平上结构和功能各异的各种分子(包括基因、蛋白质、小分子代谢物等),以及分子在生命体的内部之间、与外界环境之间的相互作用,通过精准的分析测量技术、结合计算生物学来定性、定量构建生命的数字化模型,预测生理状态或者疾病的功能、表型和转化的前沿科学。生命体的运作是在基因调控下,由许许多多的生化反应形成的复杂代谢网络。定量系统生物学将代谢网络节点的各种分子及其相互作用的反应速率数字化,构建计算模型描绘从局部到整体,最终完成整个生命活动的数字化网络图谱。在分子尺度上由基因、小分子代谢物构成的代谢网络则是定量系统生物学的关键枢纽,而小分子代谢物是这一系列事件的最终结果,它能够更准确地反映系统生命体系的瞬时状态。
随着基于液相色谱结合质谱技术的组学方法不断完善,人们对代谢网络有了更深入了解。质谱技术在检测灵敏度、色谱图分辨率和质量测量精度方面的快速提高使科学家能够一次性分析成千上万种代谢物。但是,从生物样品中获得的高分辨率质谱数据非常嘈杂,并且可能包含很多的背景信号与噪音。智能的组学大数据处理算法与工具对于代谢物的定性和定量表征至关重要。
针对基于质谱的非靶向代谢组学数据中存在的大量的假阳性与冗余的信号峰,该论文提出了色谱中心策略算法(CCS),并开发了在线互动工具来解决该领域普遍存在的问题(图2)。色谱中心策略是指通过对组学数据中提取的色谱峰形状进行数字化描述,包括对称性(Symmetry)、锯齿度(Jaggedness)、形态(Modality)、色谱质量指数(MCQ)等,并以在线互动的方式直接展示色谱峰的形态特征。同时,CPVA能够自动化注释检测峰中遇到的稳定同位素簇、加合物以及常见的782种质谱背景污染物,从而大大减少假阳性与冗余峰的数量,提高非靶向代谢组学研究的数据质量。研究团队采用受试者工作曲线(ROC)评估该算法对临床代谢组学数据的处理能力,结果显示,该算法表现出了非常出色的识别假阳性或者冗余峰的能力(AUC = 0.95)。
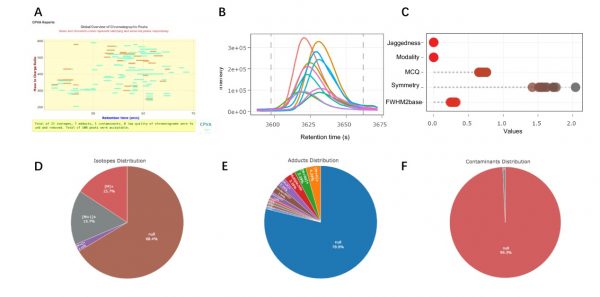
图2 CPVA算法与工具的结果展示
Bioinformatics 杂志为国际计算生物学会(ISCB)的官方期刊,由牛津大学出版社出版,是生物信息学与计算生物学的专刊、生信领域的顶尖期刊。
张文勇课题组成员、医学院兼前沿与交叉科学研究院研究助理教授栾合密为第一作者,栾合密与张文勇为论文的共同通讯作者,南科大为论文的第一作者单位。这一研究得到了国家自然科学基金委青年基金项目与深圳市教育局项目的支持。
论文链接:
https://academic.oup.com/bioinformatics/advance-article/doi/10.1093/bioinformatics/btaa200/5809525
供稿:医学院
通讯员:伍渊
编辑:苗雪宁